2025/11/21
点击次数:4237
调查和监测的数据处理
1 土壤微生物物种和功能基因多样性数据处理
土壤微生物物种和功能基因多样性监测指标包括频度(frequency)、相对频度(relative frequency)、多度(abundance)、相对多度(relative abundance)、丰度(richness)、Shannon-Wiener多样性指数、Simpson多样性指数、Pielou均匀度指数等。高通量测序数据在进行物种和功能基因多样性分析前,可以将每个样品重抽样到相同序列数,以减少因样品的测序深度不同造成的数据分析结果的误差。下面详细说明各多样性监测指标的概念和计算方法。
(1)频度 某个物种、OTU、ASV、或功能基因在调查的样本或样方内出现的频率,即某个物种、OTU、ASV或功能基因出现的样本或样方数/总样本或样方数(%)。
(2)相对频度 某个物种、OTU、ASV、或功能基因的频度占所有物种、OTU、ASV、或功能基因的频度之和的比例(%)。
(3)多度(abundance) 某个物种、OTU、ASV或功能基因在调查样本或样方出现的个体数。
(4)相对多度(relative abundance) 某个物种、OTU、ASV或功能基因的个体数占所有物种、OTU、ASV或功能基因个体数的比例(%)。
(5)丰度(richness) 在调查的样本或样方内的物种、OTU、ASV或功能基因的数量。
(6)Shannon-Wiener多样性指数 是用来描述种的个体出现的紊乱和不确定性的指标。不确定性越高,多样性也就越高,该指数能够描述两方面的信息:①种类丰度;②种类中个体分配上的均匀性。计算公式为H’ = 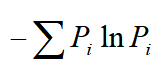 。
。
(7)Simpson 多样性指数 兼顾丰富度和均匀度,计算公式为D= 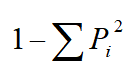 。
。
(8)Pielou均匀度指数 是指一个群落或生境中全部物种个体数目的分配状况,反映的是各个物种个体数目分配的均匀程度,计算公式为Jsw = 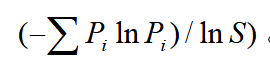 。
。
(6)—(8)式中:Pi 为物种i(功能基因i)的个体数占样地内(样品内)总个体数的比例,i=1, 2, …, S。
2 土壤微生物物种和功能基因组成数据处理
β-多样性是指不同群落之间物种或功能基因组成的相异性或物种沿环境梯度的更替速率,也被称为生境间的多样性。在扩增子测序分析中,我们普遍使用基于Unifrac距离或Bray-Curtis距离的非度量多维尺度分析(NMDS)、置换多元方差分析(PermANOVA)、主坐标分析(PCoA)和方差分解等方法检测不同生境群落之间物种或功能基因组成的相异性。Unifrac距离同时考虑了物种组成差异和系统发育关系。Bray-Curtis距离则适用于不能构建系统发育树的情况(例如真菌ITS测序、宏基因组分析,只考虑物种组成差异)。此外,unweighted(非加权)距离只考虑物种是否存在,weighted(加权)距离考虑了物种丰度的差异。如,根据OTU表和系统发育树生成Unifrac距离矩阵,绘制weighted Unifrac PCoA图是用于分析16S rRNA基因的β-多样性(Rui et al., 2015)。主坐标分析(principal coordinates analysis,PCoA)指通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值。分析对象为任意类型的距离矩阵,因此可灵活选择关联测度。PCoA是将聚类分析与主成分分析方法结合起来,用较少的主坐标对分类单元进行有效地排序,并使损失的信息最小。
3 建立数据库和数据复核
3.1 微生物多样性监测网络数据库和数据复核
土壤微生物学科的快速发展产生了大量序列数据集,目前一般存储在NCBI、ENA和MG-RAST等国际生物信息学数据库。但是这些数据库缺少用于检索、整合和重新分析多个独立数据集所必需的信息,导致现有数据集利用率低、使用时间成本高等问题。此外,微生物组数据分析涉及大量的软件和多种编程语言,学习时间成本高。因而建立整合了土壤微生物重要元数据的数据库是微生物多样性网络建设的必然要求。数据库需要同时实现数据提交和存储、数据检索和下载、比较微生物组分析、生物信息分析、实时交互式统计分析、实时交互式可视化功能。目前,中国科学院成都生物研究所建立了环境基因组云数据库,本数据库第一大特点是同时收集微生物测序数据和与之关联的环境参数信息,为用户提供数据的存储、检索和分析服务。基于测序样本详细的环境参数信息,实现了对具有特定属性的样本的精确查询,可提高数据的使用率,降低数据收集的时间成本,为微生物生态学研究提供优质的数据源。微生物组数据库不仅是一个专业的实体数据库,也是一个一站式比较群落生态学研究的云平台,为所有注册用户免费提供“数据存储—检索—生物信息学分析—统计分析和可视化”一站式服务,让用户能够获取用于meta分析的数据集,不用写代码也能进行生物信息分析、统计分析和可视化。整个平台采用JavaScript、Python、R语言开发,后台数据库使用mongoDB和Redis,HTTP服务器采用Nginx,FTP服务器采用vsftpd,所有web应用进行容器化,部署在Ubuntu 18.04环境中。目前,微生物组数据库已经收录了包括16S rRNA基因、18S rRNA基因、ITS、功能基因(nifH、phoD、pmoA等)等高质量数据集13000多套,其中包括序列文件、样本概况、空间信息、气候特征、植被信息和理化特征信息。
使用者在提交数据时将进行数据质量控制。数据质量控制包括三重把关:上传模版控制、管理员审核、脚本程序自动检测。第一,用户要按要求填写上传模版文件,从而达到数据的规范化。模版文件是一个Excel文件,由多个子表格组成,每个子表格由固定字符组成,包含了必要的土壤信息,例如经纬度、采样时间、样品描述等重要信息。第二,管理员审核用户提交的上传文件,及时纠正发现的错误。例如必填字段是否都填写完整,填写内容是否符合字段要求,数据是否符合本数据库接收条件等等。如果文件审核不通过,管理员将通知用户进行修改。最后,上传文件由脚本程序转化为数据库存储文件,如果上传文件的格式有误,则转化失败,将错误信息返回给用户进行纠正。脚本程序比管理员人工审核更适合做格式上的检查。欢迎科研工作者能够将已发表的数据集提交到微生物组数据库中存储,利用该数据库的资源解决自己的研究问题。
3.2 其他可用数据库介绍
其他可以存放的公共数据库包括:
(1)科学数据银行 科学数据银行(Science Data Bank)简称ScienceDB,是一个公共的通用型科学数据存储库,主要面向科研人员、科研项目/团队、科研期刊、科研机构及高校等利益相关者,提供科学数据汇交、长期保存、出版、共享和获取等服务,支持多种的数据获取与使用许可,在保障数据所有人权益的基础上,促进数据的可发现、可引用、可重用。ScienceDB由中国科学院计算机网络信息中心建设和维护。
(2)组学原始数据归档库 组学原始数据归档库(Genome Sequence Archive)简称GSA,是组学原始数据汇交、存储、管理与共享系统,是国内首个被国际期刊认可的组学数据发布平台。目前已整合INSDC组学数据,提供统一检索、数据下载及数据导向服务。
(3)SRA数据库 Sequence Read Archive简称SRA,隶属NCBI(National Center for Biotechnology Information),是一个保存大规模平行测序原始数据以及比对信息和元数据(metadata)的数据库。其中的数据则是通过压缩后以.sra文件格式来保存的,SRA数据库可以用于搜索和展示SRA项目数据,包括SRA主页和 Entrez system,由NCBI负责维护。
(4)ENA数据库 European Nucleotide Archive简称ENA,隶属EBI(European Bioinformatics Institute),功能等同SRA,并且对保存的数据做了注释,界面相对于SRA更友好,对于有数据需求的研究人员来说,ENA数据库最诱人的点应该是可以直接下载fastq(.gz)文件,由EBI负责维护。
4 编写监测报告及发布数据集
4.1 编写监测报告
监测报告应包括前言,监测地点概况(地理位置、植被、土壤、气候等信息),监测方法(包括工具、方法、时间等),数据处理方法,监测区域土壤微生物物种和基因多样性和组成、分布区域等,其他建议等。参考的监测报告编写格式如下。
土壤微生物监测报告编写格式
土壤微生物监测报告由封面、报告目录、正文、致谢、参考文献等组成。
1. 封面
包括报告标题、监测单位、编写单位及编写时间等。
2. 报告目录
一般列出二到三级目录。
3. 正文
包括:
(1) 前言;
(2) 监测地点概况(地理位置、植被、土壤、气候等信息等);
(3) 监测方法(工具、方法、时间等);
(4) 数据处理方法;
(5) 监测结果(监测区域土壤微生物物种和基因多样性和组成、分布区域等);
(6) 其他建议。
4. 致谢
5. 参考文献
4.2 发布数据集
每年底或次年初,发布前一年度的监测数据,数据集主要包括以下几个方面。
(1)监测样点土壤细菌、古菌和真菌的种群组成和多样性数据,包括各样点微生物在门、纲、目、科、属、OTU或ASV水平上的组成。
(2)监测样地的土壤基因组成和多样性数据,主要包括与养分循环相关的功能基因组成和丰度数据。
(3)主要森林样地大型真菌的组成和多样性,包括真菌的形态特征和图片资料。
(4)发布新分离的菌种目录及其生理特性数据。
(5)经过3年观测,发布中国典型生态系统中土壤细菌、真菌、古菌、大型真菌、土壤宏基因组和重要功能基因的组成和多样性等数据,以名录、数据集或图鉴的形成发布。
5 监测质量控制
(1)严格按照要求进行样地和样线的设置,样地设置避免在两种生态系统类型的交汇带,设置样地最好有缓冲区。
(2)根据监测目的,提前制定监测方案,收集采样地植物图鉴和大型真菌图鉴等书籍,对监测人员开展技能和安全培训,提升监测水平和安全。
(3)认真及时记录野外监测表格,注重原始数据的收集和整理,不可随意修改数据。
(4)土壤过程参数的测定过程中,尽量同一人按照相同标准完成所有样本的测定,同时设置标准样本,使不同批次或年际间的数据具有一致性。
(5)详细记录微生物测序数据分析所用的方法、软件参数和比对数据库及其版本,做到不同批次数据的分析方法相同。