2025/11/21
点击次数:4010
陆生大中型兽类监测技术规范
1 红外相机监测技术规范
1.1 监测目的
红外相机技术具有环境干扰小、可同时监测多种动物、费用相对较低、操作简单、省时省力等优势,该技术已发展成为野生动物的重要常规监测技术。应用红外相机监测技术,可开展陆生大中型兽类、地面活动鸟类等野生动物的多样性监测,从种群、群落、行为多个层面了解这些重要野生动物的现状、变化及其影响因素,为监测样区或台站的常规监测、野生动物本底调查、濒危物种保护管理提供科学依据。
1.2 适用范围
本规范规定了红外相机监测的对象、监测指标、技术要求和方法。适用于科研院所、大专院校、自然保护地、野生动物保护管理机构、生态监测专业机构等开展科学研究、资源调查、生态环境监测、生物多样性评估和保护管理成效评估等工作。
1.3 术语和定义
(1)红外相机[ infrared-triggered camera (camera trap)] 为“红外触发相机”的简称,可自动拍摄在其前面经过的动物的一种摄像装置,由相机、控制系统、传感器、存储卡等组成。
(2)有效照片(valid image) 通过相机正常拍摄而形成的,含有野生动物、人、家禽家畜等的照片或视频。如因误触发空拍、曝光过度、曝光不足等造成,缺少上述有效信息的照片或视频的则为无效照片。
(3)独立有效照片(independent image) 通常指在30 min内,某种动物(人)在同一相机位点连续出现(活动)所拍摄到的一组有效照片(视频)记为一张独立有效照片(或一次独立拍摄事件)。注意,同组中连续拍摄的照片或视频不应分别计算为有效照片。
(4)总相机日 (total camera day) 一个相机位点的相机正常连续工作24 h为一个相机有效工作日(简称相机日)。在某个抽样时段内目标保护区内所有相机位点实际工作的有效工作日的总和为总相机日。
(5)集群大小(group size) 基于红外相机图像数据,限指在同一相机位点所拍摄的一组或一张图像中同一物种的所有个体数。
(6)相对多度指数(relative abundance index,RAI) 也称为拍摄率(photographic rate)。指某一调查区域(保护区)内,每100个相机日所获取某一物种在所有相机位点的独立有效照片数。
(7)网格占有率(grid occurrence) 也被称为物种相机位点占有率。指某一调查区域内,某一物种被拍到的网格单元数或相机位点数占所有正常工作的网格单元数或相机位点数的百分率。
(8)栖息地占域率(habitat occupancy rate) 指某一物种占据研究区域(如保护区)的概率。作为评估目标物种的空间分布和栖息地相关因素之间相互关系的指标。
(9)物种丰富度(species richness) 指目标区域或群落内所记录的所有物种数目。
(10)野生动物图片指数(wildlife picture index) 简称WPI,主要用于在群落水平上调查记录物种栖息地占域率在不同时相的变化,反映大中型兽类群落的多样性变化趋势。
(11)绘制物种分布图(mapping species distribution) 通过某一物种在保护区所有监测网格的出现数据来绘制该物种的种群空间分布图、物种多度分布图等。
(12)日活动节律(diel activity rhythm) 指动物在一天24 h内的活动分布情况。动物对时间资源利用的数据是时间生态位的重要信息来源。每个物种图像所记录的具体时间可用于分析不同物种在时间轴上的活动规律,如夜行性、昼行性、晨昏活动等类型。
(13)凭证图像(voucher image) 某一物种在目标区域存在与否的凭证依据有实体标本、组织样品、痕迹和图像等。红外相机调查所获得的清晰影像资料即可作为凭证图像,是对目标区域内各物种分布记录的有效凭证依据,且每个图像均有日期时间、经纬度、海拔、植被等详细信息。
1.4 监测对象与监测内容
以陆生大中型兽类和地面活动鸟类等主要监测对象,详见表1。
表1 适用于红外相机监测的我国陆生大中型兽类和地面活动鸟类所属目科
纲 | 目 | 科 |
哺乳纲 | 长鼻目 PROBOSCIDEA | 象科 Elephantidae |
灵长目 PRIMATES | 懒猴科 Lorisidae |
猴科 Cercopithecidae |
兔形目 LAGOMORPHA | 兔科 Leporidae |
啮齿目RODENTIA | 河狸科 Castoridae |
松鼠科 Sciuridae |
豪猪科 Hystricidae |
劳亚食虫目 EULIPOTYPHLA | 猬科 Erinaceidae |
鲸偶蹄目 CETARTIODACTYLA | 骆驼科 Camelidae |
猪科 Suidae |
鼷鹿科 Tragulidae |
鹿科 Cervidae |
牛科 Bovidae |
麝科 Moschidae |
奇蹄目 PERISSODACTYLA | 马科 Equidae |
鳞甲目 PHOLIDOTA | 鲮鲤科 Manidae |
食肉目 CARNIVORA | 猫科 Felidae |
林狸科 Prionodontidae |
灵猫科 Viverridae |
獴科 Herpestidae |
犬科 Canidae |
熊科 Ursidae |
小熊猫科 Ailurida |
鼬科 Mustelidae |
鸟纲 | 鸡形目GALLIFORMES | 雉科Phasianidae |
雀形目PASSERIFORMES | 八色鸫科Pittidae |
鸦科Corvidae |
鸫科Turdidae |
鹟科Muscicapidae |
噪鹛科Leiothrichidae |
监测内容主要在物种和生态系统两个水平上进行:
(1)在物种水平,主要选择濒危物种、经济物种和指示物种等,监测其种群动态和主要影响因素;
(2)在生态系统水平,选择重要生态系统类型建立长期固定监测样地,实现对生态系统组成、结构、功能以及关键物种、濒危物种等的监测。
基于红外相机技术的兽类多样性监测指标详见表2。
表2 基于红外相机技术的兽类多样性监测指标体系
指标级别 | 指标类别 | 监测指标 | 测定方法 | 频次 |
一级指标 | 种群 | 相对多度 | 红外相机 | 1次/年 |
网格占有率 | 红外相机 |
栖息地占域率 | 红外相机 |
群落 | 物种丰富度 | 红外相机 |
多样性指数 |
栖息地 | 生境类型 | 人工调查、遥感调查 |
人类干扰 | 人工调查、红外相机、遥感调查 |
二级指标 | 个体 | 性状 | 红外相机 |
行为 |
种群 | 数量 | 红外相机 |
密度 |
种群性比 |
年龄结构 |
群落 | 种间关系 | 红外相机 |
野生动物图片指数 |
栖息地 | 植被类型 | 人工调查、遥感调查 |
地理特征 |
物候特征 | 遥感调查、红外相机 |
1.5 监测工具及技术参数要求
红外相机监测野外需要配备红外相机、卫星定位导航仪、卷尺。为保证野外监测的数据质量,建议红外相机设备至少满足以下技术参数:
(1)可拍摄照片和视频,照片分辨率不低于1200万像素,视频分辨率不低于1080 P;
(2)触发速度:PIR感应到拍照时间≤0.6 s;
(3)镜头规格:自动控制日夜型光学红外镜头,FOV≥55°;
(4)防护等级: IP66及以上;
(5)待机时间:3~6个月。
1.6 监测方法与步骤
至少需要布设60台红外相机以反映长期变化,相机之间间距不小于1 km,以减少空间自相关。森林生态系统中,通常将监测样区在地图上划分为1 km×1 km网格,对所有网格进行编号,确定监测网格的数量(图1)。如果监测样区面积较大,可采用分层抽样,根据目标区域的地形、植被、海拔和人为活动等情况设置不少于3~5个20 km2的固定监测样方。如果相机数量较少,可在不同监测网格之间对相机轮换使用。网格大小可根据生境类型、区域面积、资源(人力、财力和设备)情况进行调整,如草地、荒漠等开阔生境网格大小可适当增大。
通过GIS确定每个网格的相机预设位点(通常每个网格的中心点为预设点),导出经纬度数据,置入手持式卫星定位导航仪或其他导航软件,便于野外导航来确定每个监测网格的实际相机监测位点。在实际安装中,要综合考虑动物出现、海拔、植被和人类活动干扰强度等多种因素。通常选择兽径、水源点等动物经常活动的地方,或者动物痕迹(粪便、尿液、足迹、卧迹和遗落物等)较多的位置作为相机位点。相机前方要相对开阔,不要有草或物体遮挡,同时注意避开阳光直射以免造成较多的空拍。相机通常捆绑在树干0.3~0.5 m高的位置,具体根据目标物种高度和地形确定。安装和回收存储卡时,均需拍摄1张写有相机位点信息(安装人、相机位点编号、日期和具体时间)的“白板照片”,以记录相机是否正常工作,并为后期时间错误的校对提供依据。
工作人员根据相机位点监测信息表详细记录红外相机安放的日期、位点经纬度、周围植被类型及其他环境因子参数,有关红外相机基本性能及使用说明参考肖治术(2019)编写的《自然保护地野生动物及栖息地的调查与评估研究——广东车八岭国家级自然保护区案例分析》一书。
拓展阅读1:红外相机位点信息表
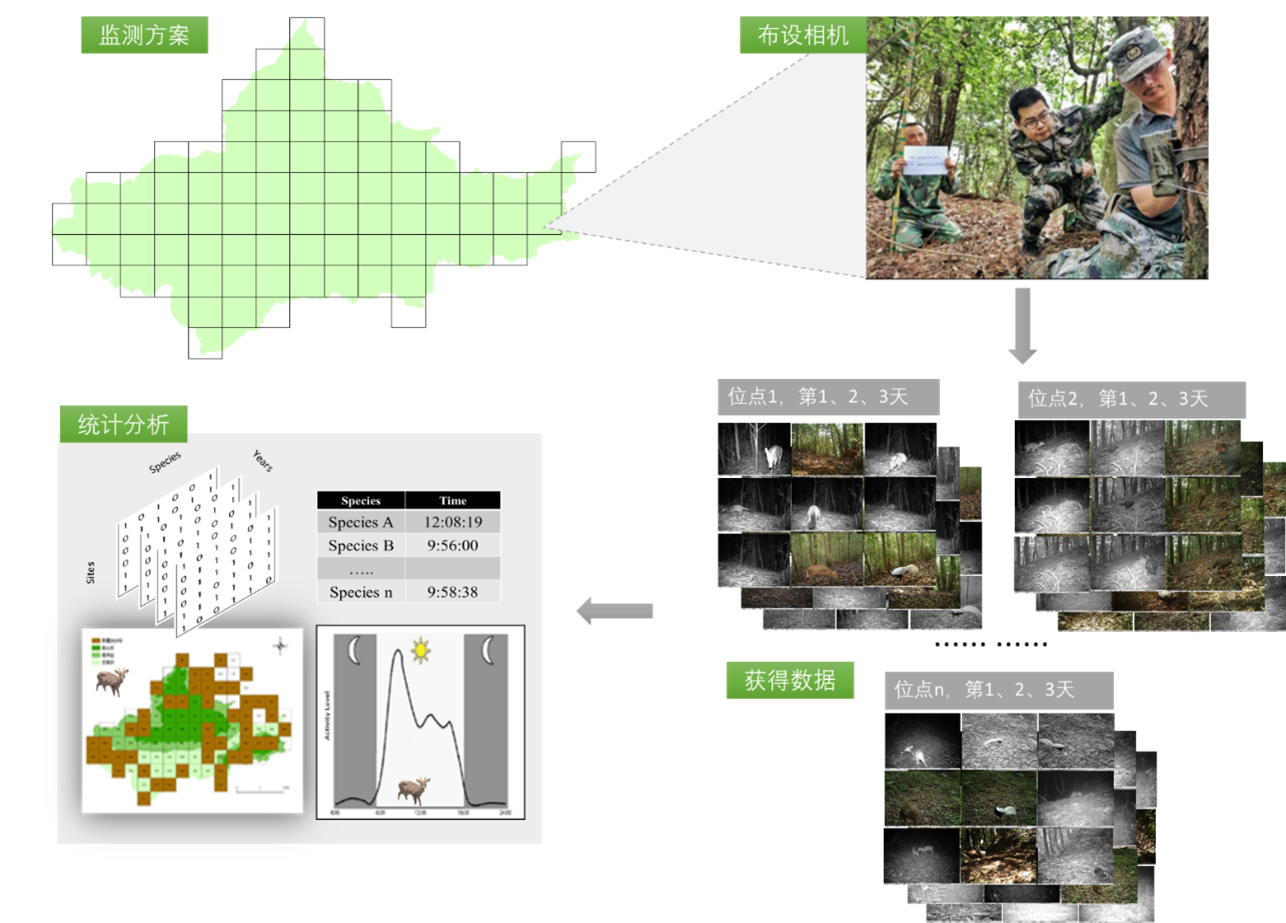
图1 红外相机监测流程
1.7 监测时间及频次
每个监测样区每年完成2~3个月的常规监测,主要在旱季(秋冬季)完成。有条件的监测样区可以进行全年监测。应尽可能在同一季节(如冬季或旱季)完成相同强度的抽样调查(如每个相机位点的调查时间为不少于60天)。
1.8 数据管理
通过上述方法所采集的图像数据和相机位点的详细信息应由专人负责管理,及时归档整理和数据备份。图像数据以“保护地/样区-相机布设年月-位点-照片记录”4级目录作为核心分级,具体项目可以根据需求在之上再添加其他的分级,例如在“监测样区”之上加上一个“项目”的级别(考虑到一个项目可以在多个保护地开展),或者在“位点”与“照片记录”之间添加一个“轮次”的级别(考虑到一个位点上可能会回收多轮数据)。具体存储结构定义如下:
监测样区编号(如车八岭):CBL
相机布设年月:202209
位点编号:CBL-相机编号A01
照片编号:IMAG0001.JPG
在图像数据采集回来后1个月内上传到野生动物监测图像数据管理系统CameraData并备份,尽量在1~2个月内完成每批数据的物种鉴定工作,导出所需数据进行统计分析和评估报告撰写等。数据使用时应有详细登记,并在论文发表时应提供数据库编号便于查询和共享利用。CameraData可完成对所上传图像的物种识别和相关数据信息的挖掘,由系统直接导出Excel数据表进行下一步数据分析,每个调查位点或网格的图像数据在系统里均有唯一的数据序列号。该系统的具体使用方法详见肖治术(2019)编写的《自然保护地野生动物及栖息地的调查与评估研究——广东车八岭国家级自然保护区案例分析》一书。
1.9 数据分析
(1) 抽样强度或者捕获强度 采用稀疏化物种累积曲线来确定最少调查时间和最小网格数量。稀疏化曲线分析指从较大样本中随机抽取部分数量的样本,并统计出这些样本中的物种丰富度,从而构建样本数(如相机日或网格数)与物种数之间的曲线。它既可以用来比较不同抽样强度的样本的物种丰富度,也可以用来验证抽样强度的合理性(Colwell & Coddington, 1994;Gotelli & Colwell, 2001)。
(2) 物种种群指标分析方法
①相对多度指数(RAI) 计算公式如下:
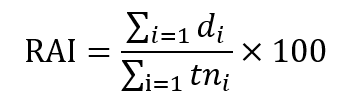
式中,tni为相机位点i的拍摄天数;di为相机位点i拍摄某一物种的独立有效照片数。
RAI主要比较每100个单位相机日内不同物种(特别是那些不能个体识别的种类)的相对密度,并假定某区域内动物的照片拍摄率与动物的密度成正相关(Carbone et al., 2001; O'Brien et al., 2003; Jenks et al., 2011)。
②网格占有率 计算公式如下:
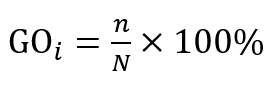
式中,n为物种i被记录到的相机位点数或网格单元数;N为所有正常工作的网格单元数或相机位点数。
③ 栖息地占域率 占域研究的主要目的是为了了解某个物种占据研究区域栖息地的比例、空间分布, 以及环境因素对物种占域状态的影响。占域模型解决了探测率常常被忽略的问题,通常用于在野外开展重复的“探测到(记为1)-未探测到(记为0)”调查,建立占域模型分析占域率。占域率作为评估目标物种的空间分布和栖息地相关因素之间相互关系的指标。数值为0~1,0表示没有分布,1表示在所有调查点均有分布。通常表示为:
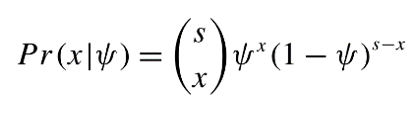
式中,Ψ为样本单元被占据的概率,χ为被占据的样本单元数量,s为样本单元数量。具体分析参见附录。
④日活动节律 红外相机影像中的物种和拍摄时间等信息可以为分析动物时间尺度的活动节律提供有效信息,目前应用较多的为日活动节律分析,通常采用核密度估计方法。具体分析参见附录。
(3) 群落特征分析方法
①日活动节律重叠度 运用核密度分析方法,还可以计算两种物种日活动节律的重叠系数,用于揭示群落中同域分布物种时间资源利用的差异及共存机制。
②多物种占域 在不完全探测存在情况下,采用多物种占域(multispecies occupancy)模型进行物种丰富度阈值评估。该方法基于对物种水平占域模型的集成,通过多次重复的调查收集探测到/未探测到的物种信息,将各物种水平模型的参数视为随机效应,与群落内物种间变异的先验分布相关(Kéry & Royle, 2008)。该模型分3层,涉及物种(i)、位点(j)和重复调查(k)。第一层代表真实的出现状态(w)包括群落中所有观测到和未观测到的物种,取决于物种是群落中一员的概率Ω(公式3-1)。第二层是用于解译部分观测到物种的出现状态(z)这一生态学过程,与每个物种的真实出现状态及其每个位点的占域率有关(公式3-2)。第三层用于解释探测过程,即基于重复调查的探测历史(y),依赖于观测到各个物种的出现状态及其每个位点每次重复调查的探测率(公式3-3)。
wi ~Bernoulli(Ω) (3-1)
zi,j|wi ~Bernoulli(wi*ψi,j) (3-2)
yi,j,k|zi,j~ Bernoulli(zi,j*pi,j,k) (3-3)
通过考虑不完全探测和数据扩增,可以解释群落中未观测到的部分(Dorazio & Royle, 2005)。该模型通过在物种间借用信息而促进估计,从而促进共享参数的估计,并且可以对稀有物种模拟物种水平的影响。同时估计栖息地和景观变量对群落结构和物种丰富度的影响(Kéry & Royle, 2009; Russell et al., 2009)
③野生动物图片指数(WPI) WPI是O’Brien等(2010)所提出的用于估计大中型动物相对丰富度的综合指标。WPI计算主要基于红外相机监测数据,应用占域模型估计群落中每个物种在一段时间内占域率,如调查点k在j时间的野生动物图片指数为k调查点上所有n个物种在j时间占域率与调查起始时间所有物种占域率的比值的几何平均值(TEAM采用调查起始年为计算WPI的参考基线,Beaudrot et al., 2016)。
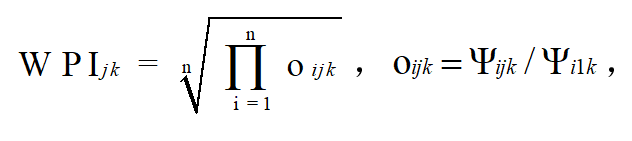
式中,为调查点k的物种i在时间j的相对占域率;为调查点k的物种i在j时间的占域率;为调查点k的物种i在调查起始时间的占域率。
2 粪便DNA调查分析技术规范
2.1 监测目的
非损伤性分析材料包括粪便、毛发、尿液、动物咀嚼过的残渣上所残留的唾液、羽毛、卵壳等(Beja-Pereira et al., 2009),其中粪便是最容易获取的分析样品。通过对粪便中目标动物的DNA提取,再进行相关的遗传学分析,可以对目标动物进行物种、个体和性别的鉴定,同时还可进行目标动物的种群数量估计以及遗传结构、遗传多样性等遗传管理方面的分析(Kleemann et al., 2021; Kohn, 2010; Mondol et al., 2009; Ottewell et al., 2020)(图2)。目前该技术已成为野生动物管理方面一项重要的监测技术,并可为野生动物的监测和保护管理提供科学依据。
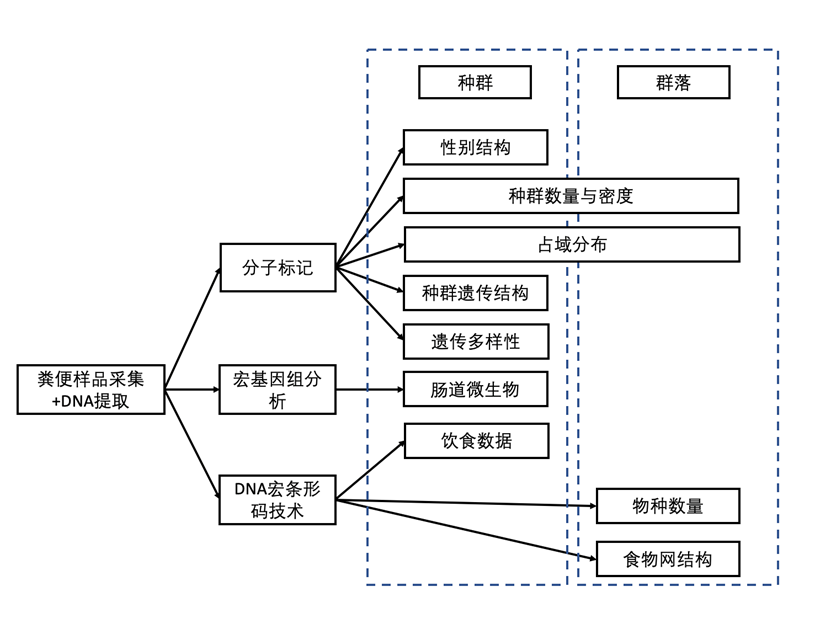
图2 粪便DNA应用研究领域
2.2 适用范围
本规范规定了动物粪便样品的采集、保存、DNA提取及质量鉴定、基因型鉴定、物种鉴定、个体鉴定、性别鉴定以及种群遗传结构、遗传多样性分析和种群数量估计的鉴定程序与要求。适用于对兽类有进行非损伤性取样和DNA调查分析需求的研究。
2.3 术语与定义
(1)非损伤性遗传取样(noninvasive genetic sampling,NGS) 在不接触或者伤害野生动物的情况下,通过收集动物脱落的毛发或羽毛、粪便、尿液、食物残渣(含有口腔脱落细胞)、角、鳞和卵壳等不同形式的样品而进行遗传学分析的一种取样方法。
(2)定量PCR(quantitative PCR,qPCR) 通常指实时荧光PCR(quantitative real-time PCR)。以荧光化学物质实时检测每次聚合酶链式反应(PCR)循环后产物总量,对待测样品中的特定DNA序列进行定量分析的方法。
(3)微卫星DNA(microsatellite DNA) 又称短串联重复序列或简单重复序列(short tandem repeats, STRs),是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成。具有数量多、在基因组内分布均匀、多态性信息丰富、易于检测等优点被作为优良的遗传标记(genetic marker)而得到广泛应用。
(4)微卫星PCR 对微卫星DNA进行PCR扩增。
(5)引物(primer) 引物是根据目标扩增区域DNA序列人工合成的寡核苷酸序列,在PCR过程中与模板DNA特异结合起始DNA合成的大分子。
(6)基因型(genotype) 某一生物个体全部基因组合的总称。它反映生物体的遗传构成,即从双亲获得的全部基因的总和。遗传学中具体使用的基因型,往往是指某一性状的基因型。
(7)等位基因(allele) 位于一个同源染色体相同位置上控制同一性状不同序列的基因。
(8)杂合子(heterozygote) 同源染色体同一位点上的两个等位基因不相同的基因型个体。
(9)纯合子(homozygote) 二倍体中同源染色体上相同位点等位基因相同的基因型个体。
(10)杂合度(heterozygosity) 称群体的平均杂合性或杂合度,它是群体遗传变异的一个度量参数,是指某一基因座上的等位基因是杂合体的频率。
(11)哈迪-温伯格平衡(Hardy-Weinberg law) 是种群遗传学中最重要的原理,是指在一个不发生突变、迁移和选择的无限大的随机交配种群中,种群的基因频率和基因型频率将逐代保持平衡稳定不变。
(12)DNA宏条形码技术(DNA metabarcoding) DNA 宏条形码技术是指整合DNA 条形码和高通量测序技术,利用高通量测序手段对从环境样本中(如土壤、水和粪便等)提取的总 DNA 和降解 DNA 进行高通量多物种鉴定的技术,为动物食性的分子鉴定提供了新的机遇。
2.4 监测对象与监测内容
兽类为主要监测对象,在个体和种群两个水平上进行:①在个体水平,主要进行物种、个体以及性别的鉴定;②在种群水平,进行种群数量估计以及遗传结构、遗传多样性等遗传管理方面的分析。基于粪便DNA技术的兽类多样性监测指标详见表3。
表3 粪便DNA监测指标体系
指标级别 | 指标类别 | 监测指标 | 测定方法 | 频次 |
二级指标 | 物种 | 性别 | 粪便DNA法 | 1次/年 |
食性结构 |
种群 | 数量 |
密度 |
种群性比 |
年龄结构 |
遗传结构 |
遗传多样性 |
2.5 监测方法与步骤
2.5.1样品采集
采用非损伤性遗传取样法进行粪便样品采集。原则上只采集兽类短期内排出的粪便,用卫星定位导航仪对每一个样品的地理位置进行定位,避免采同一个体一次排便样本,样品采集间隔50 m以上。为避免交叉污染,取样时均佩戴一次性无菌手套和口罩,每个样品放入一个洁净的采样管,通过酒精浸泡或硅胶方式室温下保存。
2.5.2主要试剂
粪便DNA提取试剂盒(Qiagen,Germany)、蛋白酶K(上海生工生物工程有限公司)、无水乙醇、缓冲液、细胞裂解液(10 mmol /L Tris-HCl溶液 pH 8.0,0.1 mol/L EDTA溶液 pH 8.0,0.2 mol/L NaCl溶液,5 g/L SDS溶液)、Taq酶(Takara)等。
2.5.3主要仪器
水浴锅(TE-10D,TECHNE),高速冷冻离心机(1-15K,GMA),PCR仪(ABI-9700,ABI),振荡器(2002型,常州国华电器有限公司),垂直电泳槽(L222887,西域)。
2.5.4粪便基因组DNA的提取
采用QIAamp DNA Stool Kit (Qiagen,Germany)来提取粪便样品中基因组DNA。为了监测实验过程中的污染情况,每提取10个粪便样品DNA即设置两个空白对照。粪便DNA提取的具体操作如下:
(1)称取约100 mg粪便样品放入2 mL离心管中;
(2)加入1.6 mL ASL缓冲液,充分振荡,然后25℃ 900 r/min振荡过夜;
(3)12 000 r/min离心5 min,转移上层液体1.4 mL至新的2 mL离心管中;
(4)加入一片Inhibit EX,充分振荡,直到药片完全溶解;
(5)室温静置5~10 min,直至看到分层;
(6)12 000 r/min离心10 min,转移上层悬浊液至新的1.5 mL离心管中,继续全速离心6 min;
(7)取25 uL蛋白酶K至新的2 mL离心管中,将步骤(6)中的上清液转移至该管中,振荡混匀;
(8)加入600 uL AL缓冲液,立即振荡混匀15 s,70 ℃孵育10 min;
(9)加入600 uL 无水乙醇,充分振荡;
(10)小心转移600 uL步骤(9)中的混合液至QIAamp DNA吸附柱中,全速离心2 min;
(11)换一个新的吸附柱,弃离心液;
(12)将步骤(9)剩下的混合液按步骤(10)重复,直至所有的混合液均离心完毕;
(13)加入500 uL buffer AW1溶液到吸附柱中,全速离心2 min,弃离心液;
(14)加入500 uL buffer AW2,全速离心5 min,弃离心液;
(15)将吸附柱转移到1.5 mL离心管中,加入200 uL buffer AE;
(16)室温下孵育1 h,全速离心2 min;
(17)分装DNA,取出100 uL,分装到0.5 mL的离心管中,每管20 uL,4℃保存,直至整个PCR实验完成;其余100 uL DNA样品-20 ℃保存。
2.5.5物种和基因型鉴定
2.5.5.1样品基因组DNA质量鉴定
通过定量PCR方法来测定样品中模板DNA的含量(Morin et al., 2001)。
2.5.5.2微卫星位点的筛选和基因型的确定
(1)微卫星位点的筛选
①微卫星位点的筛选 采用三碱基(trinucleotide)或者四碱基(tetranucleotide)重复的微卫星位点在扩增的时候能够极大地减少非特异性产物,降低基因型分型错误的几率。因此建议采用三碱基或者四碱基重复的微卫星位点(Edwards et al., 1991; Bradley et al., 2000)。
②微卫星PCR 为了克服常规方法扩增粪便样品DNA过程中出现极低的扩增成功率和较高的基因型分型错误,获得可信的基因型,Arandjelovic 等 (2009) 发展了two-step multiplex PCR 方法。相比传统的PCR方法,该方法能够显著的提高PCR扩增成功率,同时降低等位基因缺失率(dropout rate),即使样品中含有极低量的模板DNA(﹤25 pg/uL)也能够扩增得到可靠的基因型。
a. PCR反应体系 根据物种选择合适的反应体系,一般应包括: SuperTaq buffer (HT Biotechnology), Mg2+,上下游引物,dNTP各组分,BSA,SuperTaq (HT Biotechnology) premixed 2:1 with Taq Start Antibody (BD Biosciences),以及模板 DNA等。
b.PCR参数如下 根据引物的结合特点,进行退火温度、循环次数和延伸时长的选择,进行PCR扩增。
c.第二步Singleplex PCRs条件如上,需要做出略微的改动:采用适量 1:100 稀释的上述 multiplex PCR产物作为第二步singleplex PCR的模板,并且将Mg2+的用量减半,同时减少酶以及 SuperTaq (HT Biotechnology) premixed 2:1 和 TaqStart Antibody的使用量。每一次 singleplex PCR仅包含一对微卫星引物:采用FAM、HEX和 NED 等荧光标记的正向引物和反向引物。
d.PCR条件跟第一步multiplex PCR相似,只是将退火温度设定为待扩增的微卫星引物的特定退火温度。PCR结束后,取出扩增产物在2.5% 的琼脂糖凝胶上进行电泳检测。
e.将扩增成功的微卫星 PCR 产物在 ABI PRISM 3100 Genetic Analyser上进行基因扫描,利用 GeneMapper Software version 3.7 (Applied Biosystems) 对片段大小进行判读。每一个粪便样品扩增3次,每一个96孔板设置6个阴性对照。
以上流程均为普遍性规范,具体反应体系、反应参数以及各试剂用量等需要根据物种以及引物特性而定。
(2)物种鉴定 纯化后的PCR产物使用ABI PRISM3730xl测序仪(Applied Biosystems, Foster City, California)进行测序。测序结果通过软件Chromas v2.22 (Technelysium Pry Ltd., Australia)处理, 得到长度约320 bp的单一序列。然后用NCBI (National Center for Biotechnology Information)的BLAST程序检索测序片段在其数据库GenBank中的匹配序列。最佳匹配序列与待测序列的覆盖度(coverage)不低于95%, 一致度(identity)大于95%时认为检索结果有效。结合当地动物分布信息, 对序列进行筛选, 当覆盖度达到100%、一致度达到98%及以上, 且对应物种可能分布在研究地点时, 认为粪便样品来自匹配序列对应的物种。若存在不止一种物种的匹配序列, 则根据研究地点本底信息及物种的分布特征排除不符合的物种(Xiong et al., 2016)。
(3)基因型的确定 如果两次或两次以上PCR中都出现相同的杂合子,那么认为该位点为杂合子。否则,重新进行三次PCR扩增,结合第一遍扩增结果,如果出现两次以上的两个等位基因则记录为杂合子。根据样品中模板DNA的含量,如果三次或以上的PCR反应均为相同的纯合子,认为该位点为纯合子。
2.5.6个体性别鉴定
随着分子生态学和保护遗传学的发展,基于非损伤性遗传取样的分子性别鉴定技术被广泛运用于动物个体的性别鉴定。目前最常用的分子性别鉴定技术,选用不同引物对不同长度的同源X-Y染色体序列进行PCR扩增,通过电泳对扩增产物进行可视化来判断个体的性别,其中纯合子(XX)为雌性,杂合子(XY)为雄性(Villesen & Fredsted, 2006)。具体步骤如下:
从相关文献中筛选出一对能够辨别目标物种个体性别的微卫星引物(Inoue et al., 2016; Matsunaga et al., 2014; Villesen & Fredsted, 2006)。通过引物进行PCR扩增得到长度不同的X和Y染色体序列。由于只有雄性个体具有Y染色体,因此,如果通过凝胶电泳检测到两条不同的条带,那就表明该样品来源于雄性个体,若仅检测到一条条带(即X片段),则认为该个体为雌性。与微卫星PCR类似,根据所鉴定的物种选择合适的反应体系,根据引物的结合特点,选择合适的退火温度、循环次数和延伸时长。PCR的过程依次为:变性-退火-延伸-循环-延伸。每个样品重复3次PCR,PCR扩增产物在2.5% 的琼脂糖凝胶上电泳1 h(100 V)进行检测。3次扩增均出现X条带和Y条带的样品判定为雄性,只出现X条带的样品被判定为雌性。
以上流程均为普遍性规范,具体反应体系、反应参数以及各试剂用量等需要根据物种以及引物特性而定。
2.7 监测时间及频次
繁殖季节,一年一次。
2.8 数据管理
可开发数据共享平台或者网站进行数据管理。
2.9 数据分析
2.9.1样品或标本保存
兽类粪便样品通过酒精或硅胶方式在室温下保存。
2.9.2调查结果整理
(1)个体鉴定 采用CERVUS 3.0软件 (Kalinowski et al., 2007) 来寻找数据中相匹配的基因型。判断不同粪便样品来自于同一个个体的原则是:①所有的微卫星位点的基因型均相同;②只有一个位点上的一个等位基因存在差异(Bellemain et al., 2005; Solberg et al., 2006)。为了确定所选择的微卫星位点数量是否达到了个体鉴定的要求,采用CERVUS 3.0(Kalinowski et al., 2007)软件计算位点的 PIDsib 和 PID,它们分别指全同胞个体之间具有相同基因型的概率和无亲缘关系个体之间具有相同基因型的概率。我们采用DROPOUT 和 MICRO-CHECKER 软件来检测数据中可能存在的无效等位基因及等位基因缺失等现象。
(2)种群遗传多样性分析 采用 CERVUS 3.0软件来计算每个位点的等位基因数(A)、期望杂合度 (HE)、观察杂合度 (HO) 以及检测每个位点是否符合哈-温平衡(Hardy-Weinberg equilibrium)。采用FSTAT 2.9.3.2软件(Goudet, 2001)来检测位点间是否存在连锁不平衡 (linkage disequilibrium)。
(3)种群数量估计 如果样品采集强度足够大的话,可能会在研究区域内的不同地方采集到同一个个体的不同粪便样品。如果把其中任何一次采集到的粪便样品作为一次标记,那在其他地方采集到的该个体粪便样品就可以看作是对该个体的重捕。为此可将所采集到的所有粪便样品归为一个取样周期,然后采用三种方法来估计种群数量。Miller 等(2005)提出了采用最大似然法(maximum likelihood)来估计种群数量的方法,该方法在软件CAPWIRE中运行。由于可能在一次取样过程中采集到同一个体的多个粪便样品,因此将在同一时间同一地点采集到的同一个体的不同粪便样品看作是一次标记,以避免所谓的“假重捕”。采用软件中的ECM(even capture probability model)及TIRM (the two innate rates model) 模型来估计种群数量。ECM模型假定不同的个体具有相同的捕捉概率,而TIRM模型则允许个体之间的捕捉概率有差异。由于不知数据中个体之间是否存在捕捉概率的差异,因此上述两种方法都可被采用。通过运行10 000次的bootstrap检验来产生两种模型下的种群数量及95% 水平上的置信区间。作为比较,也可采用Mh-Chao方法来估计种群数量。该方法在实际种群数量大于100的时候能够得到较好的估计值。该方法在CAPTURE软件中运行,同样产生一个种群数量的估计值及95% 水平上的置信区间。上述3种方法均假设种群内没有出生和死亡、没有迁入和迁出,是一个封闭群体。
参考文献
1. 国家林业局调查规划设计院,中国科学院动物研究所. 全国陆生野生动物资源调查自动相机法调查技术细则(试行)[Z].2017.
2. 李明,魏辅文,绕刚,等. 非损伤性取样法在保护遗传学研究中的应用[J]. 动物学报,2001, 47:338-342.
3. 肖治术, 李欣海, 王学志, 等. 探讨我国森林野生动物红外相机监测规范[J]. 生物多样性, 2014, 22(6): 704-711.
4. 肖治术, 李欣海, 王学志. 野生动物多样性监测图像数据管理系统CameraData介绍[J]. 生物多样性, 2014, 22(6): 712-716.
5. 肖治术. 自然保护地野生动物及栖息地综合调查与评估研究——广东车八岭国家级自然保护区案例研究[M]. 北京:中国林业出版社, 2019。
6. Arandjelovic M, Guschanski K, Schubert G, et al. Two-step multiplex polymerase chain reaction improves the speed and accuracy of genotyping using DNA from noninvasive and museum samples [J]. Molecular Ecology Resources, 2009, 9: 28-36.
7. Beja-Pereira A, Oliveira R, Alves PC, Schwartz MK, Luikart G. Advancing ecological understandings through technological transformations in noninvasive genetics [J]. Molecular Ecology Resources, 2009, 9:1279-1301.
8. Bellemain E, Swenson JE, Tallmon D, et al. Estimating population size of elusive animals with DNA from hunter-collected feces: four methods for brown bears [J]. Conservation Biology, 2005, 19:150-161.
9. Bradley BJ, Boesch C, Vigilant L. Identification and redesign of human microsatellite markers for genotyping wild chimpanzee (Pan troglodytes verus) and gorilla (Gorilla gorilla gorilla) DNA from faeces [J]. Conservation Genetics, 2000, 1: 289-292.
10. Edwards A, Civitello A, Hammond HA, et al. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats [J]. American Journal of Human Genetics, 1991, 49: 746–757.
11. Goudet J. FSTAT, a program to estimate and test gene diversities and fixation indices (version 2.9.3.2) [CP]. 2001.
12. Inoue E, Ogata M, Seino S, et al. Sex identification and efficient microsatellite genotyping using fecal DNA in proboscis monkeys (Nasalis larvatus) [J]. Mammal Study, 2016, 41(3)141-148.
13. Kalinowski ST, Taper ML, Marshall TC. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment [J]. Molecular Ecology, 2007, 16: 1099-1106.
14. Kleemann S, Sandow D, Stevens M, et al. Non-invasive monitoring and reintroduction biology of the brush-tailed rock-wallaby (Petrogale penicillate) in the Grampians National Park, Australia [J]. Australian Journal of Zoology, 2021, 69 (2): 41-54.
15. Kohn MH. Noninvasive genome sampling in chimpanzees [J]. Molecular Ecology, 2010, 19: 5328-5331.
16. Lee JH, Park Y, Choi J R, et al. Comparisons of three automated systems for genomic DNA extraction in a clinical diagnostic laboratory [J]. Yonsei Medical Journal, 2010, 51:104-110.
17. MacKenzie DI. Occupancy estimation and modeling: inferring patterns and dynamics of species occurrence [M]. Salt Lake City: Academic Press, 2006.
18. Matsunaga T, Ieda R, Hosoya S, et al. An efficient molecular technique for sexing tiger pufferfish (fugu) and the occurrence of sex reversal in a hatchery population [J]. Fisheries Science, 2014, 80(5): 933-942.
19. Meredith M, Ridout M. Overlap: estimates of coefficient of overlapping for animal activity patterns [CP]. R package version 0.2, 4, 2014.
20. Miller CR, Joyce P, Waits LP. A new method for estimating the size of small populations from genetic mark-recapture data [J]. Molecular Ecology, 2005, 14: 1991-2005.
21. Mondol S, Karanth KU, Kumar NS, et al. Evaluation of non-invasive genetic sampling methods for estimating tiger population size [J]. Biological Conservation, 2009, 142(10): 2350-2360.
22. Morin PA, Chambers KE, Boesch C, et al. Quantitative PCR analysis of DNA from noninvasive samples for accurate microsatellite genotyping of wild chimpanzees (Pan troglodytes verus) [J]. Molecular Ecology, 2001, 13: 2089-2094.
23. Ottewell K, Thavornkanlapachai R, McArthur S, et al. Development and optimization of molecular assays for microsatellite genotyping and molecular sexing of non-invasive samples of the ghost bat, Macroderma gigas [J]. Molecular Biology Reports, 2020, 47 (7): 5635-5641.
24. Pan D, Li Y, Hu HX, et al. Microsatellite polymorphisms of Sichuan golden monkeys [J]. Chinese Science Bulletin, 2005, 50:2850-2855.
25. Potter S, Eldridge MDB, Cooper SJB, et al. Habitat connectivity, more than species’ biology, influences genetic differentiation in a habitat specialist, the short-eared rock-wallaby (Petrogale brachyotis) [J]. Conservation Genetics, 2012, 13:937-952.
26. Rowcliffe M. Activity: animal activity statistics [CP]. R package version 1.1, 2016.
27. Solberg KH, Bellemain E, Drageset OM, et al. An evaluation of field and noninvasive genetic methods to estimate brown bear (Ursus arctos) population size [J]. Biological Conservation, 2006, 128:158-168.
28. Taberlet P, Waits LP, Luikart G. Noninvasive genetic sampling: look before you leap [J]. Trends in Ecology and Evolution, 1999, 14:323-327.
29. TEAM Network. Terrestrial vertebrate (camera trap) protocol implementation manual, v. 3.0 [CP]. Tropical ecology, assessment and monitoring network, Center for Applied Biodiversity Science, Conservation International, Arlington, VA, USA, 2008.
30. Villesen P, Fredsted T. A new sex identification tool: one primer pair can reliably sex ape and monkey DNA samples [J]. Conservation Genetics, 2006, 7:455-459.
31. Xiong MY, Shao XN, Long Y, et al. Molecular analysis of vertebrates and plants in scats of leopard cats (Prionailurus bengalensis) in southwest China [J]. Journal of Mammalogy, 2016, 97: 1054-1064.
32. Yu SC, Wang Y, Li X, et al. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 [J]. Optik, 2017, 145: 555-560.